Лекции

1
Лекция 1. Подготовка библиотеки
01:24:24
2
Семинар 1. Базовая работа с прочтениями
01:47:41
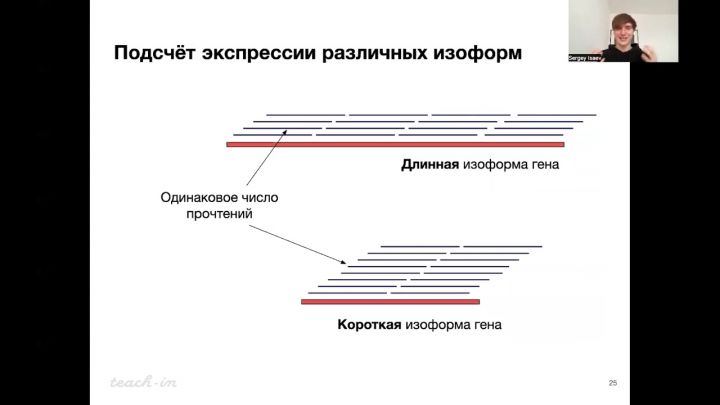
3
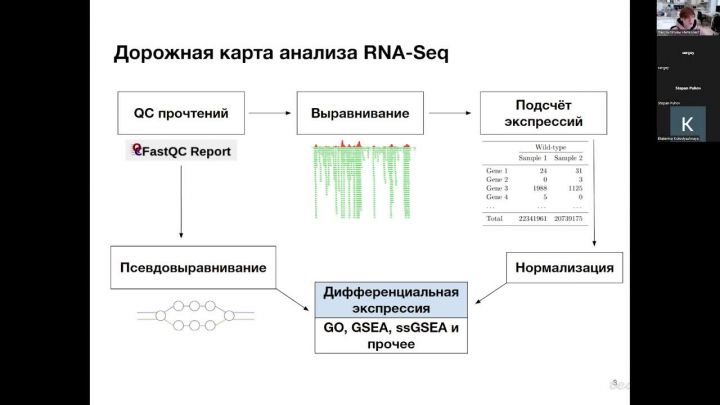
Лекция 2. Выравнивания и псевдовыравнивания. Подсчёт экспрессии
01:46:54
4
Семинар 2. ЕМ-алгоритм и kallisto
00:58:41
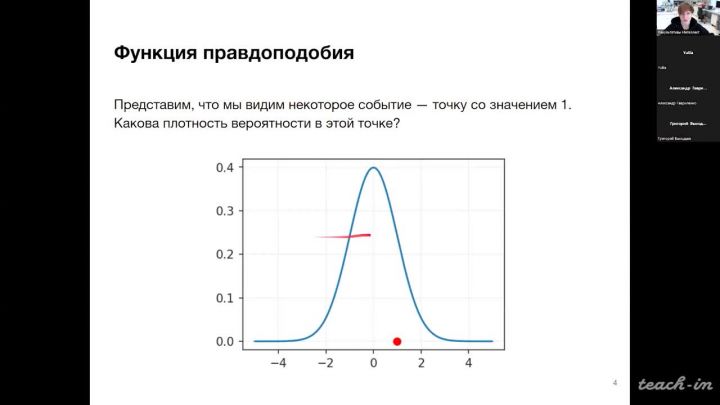
5
Лекция 3. Распределения в омиках. Методы нормализации
01:51:43
6
Семинар 3. Определение максимально правдоподобных распределений для данных
00:59:32
7
Лекция 4. Дифференциальная экспрессия
01:24:06
8
Семинар 4. Определение дифференциально экспрессированных генов. Работа с пакетами DESeq2 и edgeR
01:36:36
9
Лекция 5. Функциональный анализ
01:32:39
10
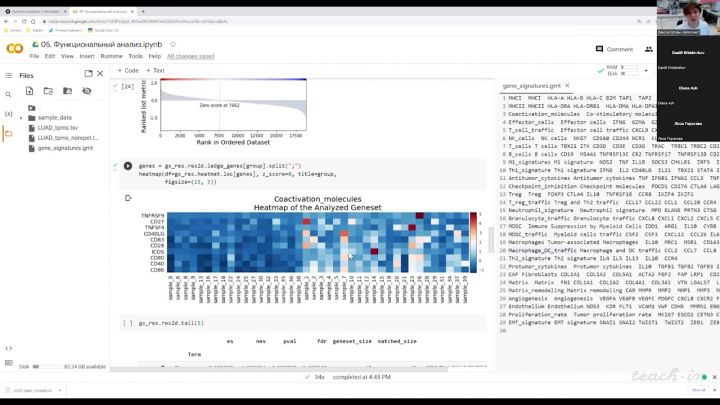
Семинар 5. Функциональный анализ RNA-Seq
00:55:06
11
Лекция 6. Транскриптомика одиночных клеток
01:34:41
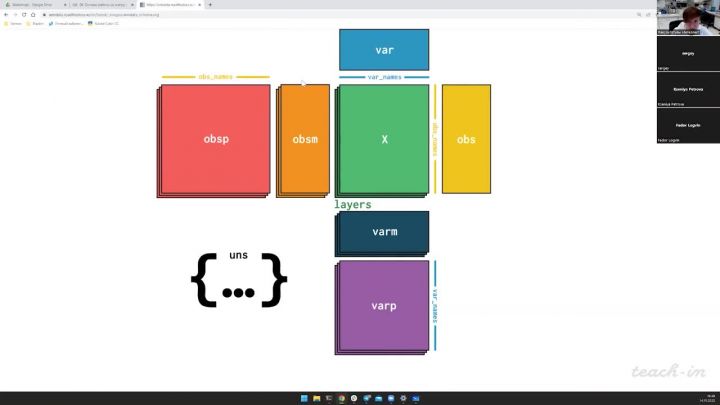
12
Семинар 6. Основы работы с библиотеками scanpy и Seurat
00:57:34
13
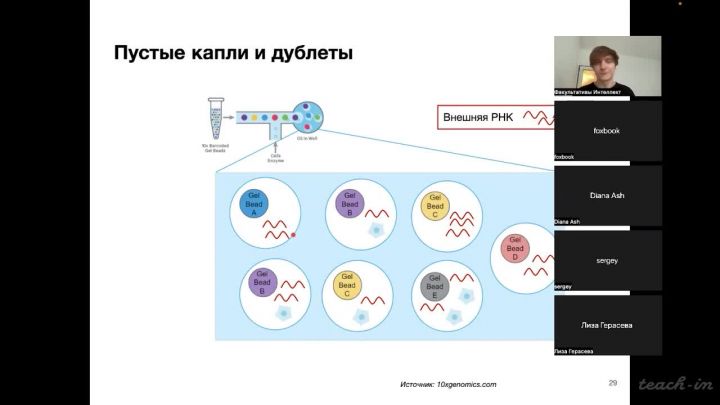
Лекция 7. Контроль качества клеток в scRNA-Seq
01:24:18
14
Семинар 7. QC
01:01:46
15
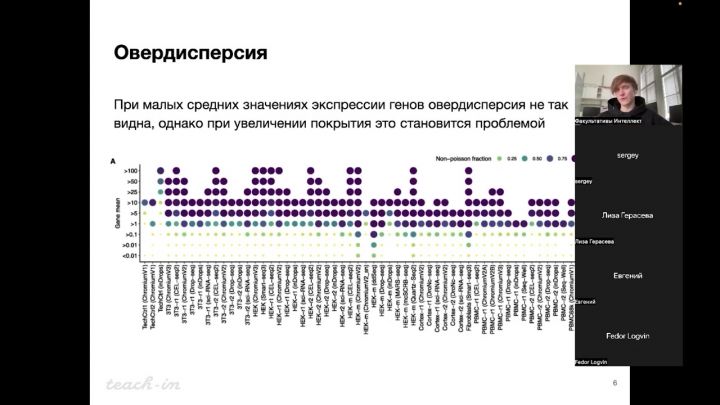
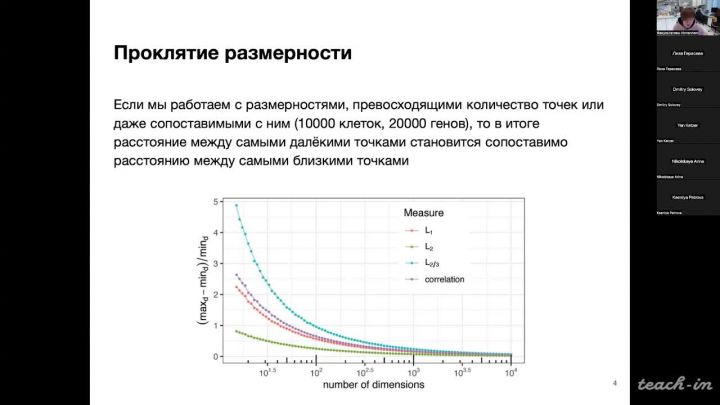
Лекция 8. Контроль за дисперсией
01:31:45
16
Семинар 8. Контроль за дисперсией данных в scRNA-Seq
01:31:19
17
Лекция 9. Методы снижения размерности
01:50:55
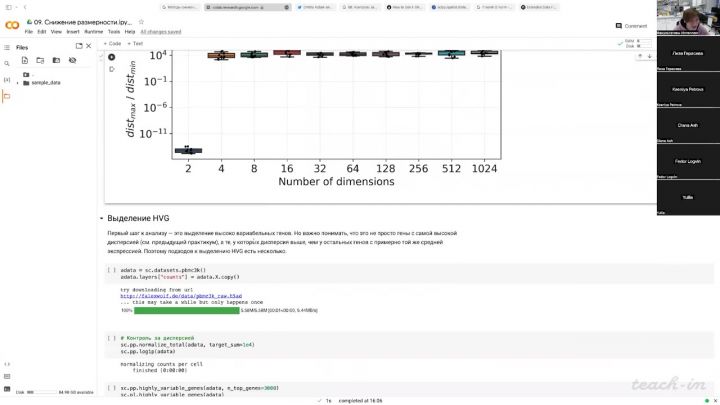
18
Семинар 9. Методы снижения размерности
01:04:38
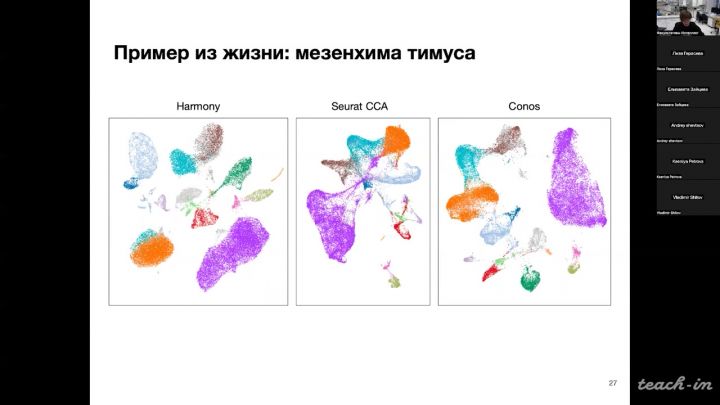
19
Лекция 10. Коррекция батч-эффекта
01:36:32
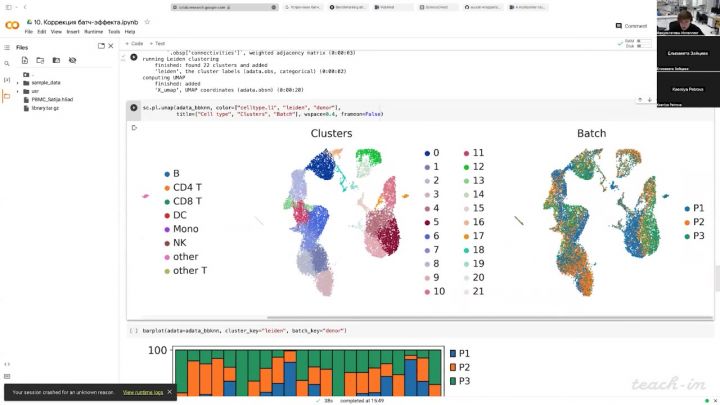
20
Семинар 10. Коррекция батч-эффекта
00:50:55
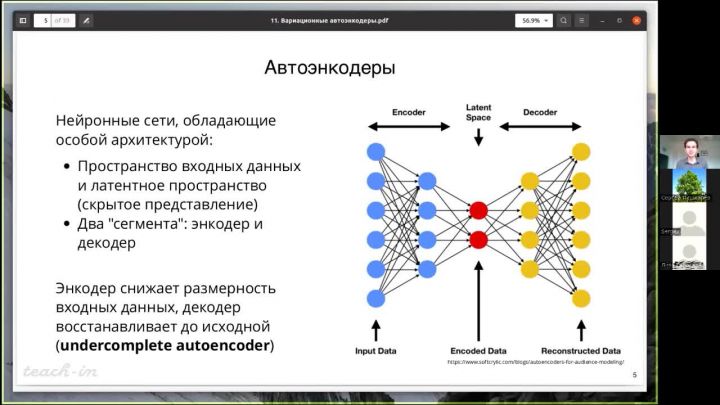
21
Лекция 11. Использование вариационных автоэнкодеров для процессинга scRNA-seq. scVI-tools
01:29:16
22
Семинар 11. Автоэнкодеры на PyTorch. Препарирование scVI
01:33:26
23
Лекция 12. Кластеризация
00:50:34
24
Семинар 12. Кластеризация и дифференциальная экспрессия
01:27:04
25
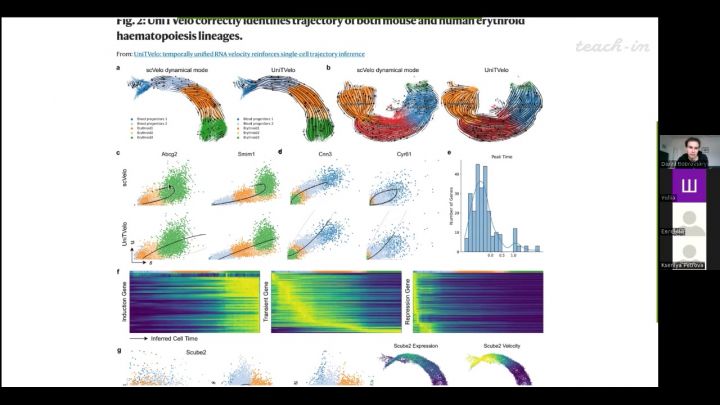
Лекция 13. Определение траекторий дифференцировки клеток в scRNA-seq
02:01:13
26
Семинар 13. Дифференцировка клеток. Определение генов, меняющих свою экспрессию по ходу псевдовремени
01:17:38
27
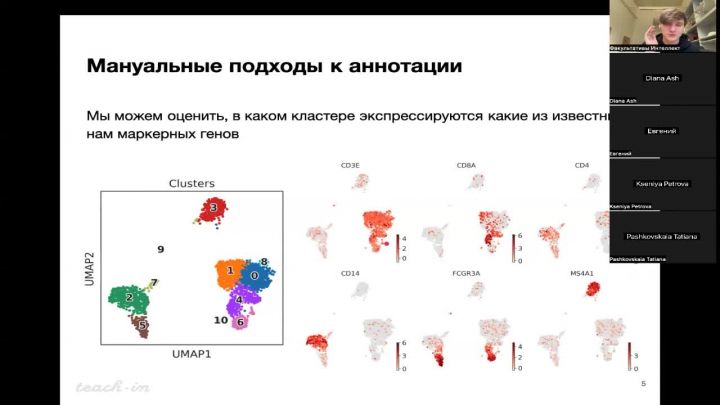
Лекция 14. Определение типов клеток
01:19:27
28
Семинар 14. Определение типов клеток
00:46:46
29
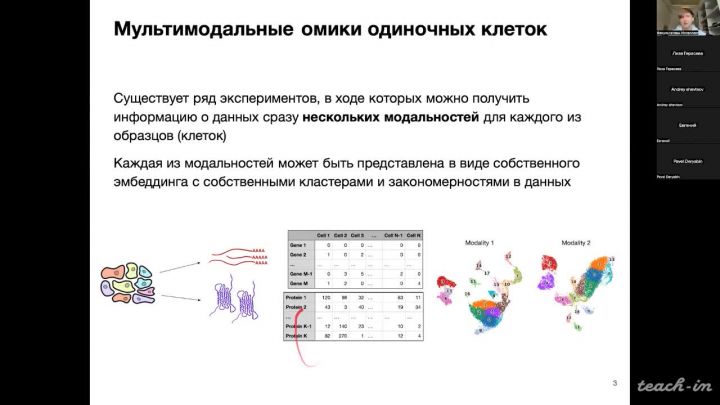
Лекция 15. Анализ мультимодальных омик одиночных клеток
01:26:22
30
Семинар 15. Анализ CITE-Seq
00:54:33


